发布日期:2018-11-08 10:02 来源:活性炭网 作者:活性炭网 浏览数:
抽象 使用PCR扩增的16S rRNA基因片段的Illumina MiSeq分析研究在St.Paul地区水服务(SPRWS)操作的全规模生物活性颗粒活性炭(BAC)过滤器的细菌群落组成。 在12个月的研究期间,这些细菌群
公共自来水公司使用地表水为居住在美国的近70%的人们生产高质量的饮用水(1)。处理地表水的常规方法涉及一系列单元操作,包括凝结,絮凝,沉降,过滤和消毒(2)。这些技术的应用已被普遍证明是有效的保护公众健康(2),尽管许多例外已报告(3)。鉴于公共供水的推定安全性,水消费者越来越关注公共供水的味道和气味以及其他美学问题(4)。
公共水公用事业,因此,已经开始增加他们的处理过程以特异性除去调味和产生气味的化合物,例如土臭味素和2-甲基异莰醇(5,- 7)。虽然一些替代技术都可以使用,使用过滤的生物活性,颗粒活性炭(BAC)已被证明,因为它的简单性(即,它很容易加装到预先存在的操作)和有效性的成功(8,9)。过滤与BAC从与由于其高吸附容量的常规粒状介质(例如,砂和无烟煤)过滤不同(6,8),以及一种生长在其表面上的活性生物膜(10,- 13)。所述BAC过滤介质上的生物膜通过代谢最初吸附土臭味素提供溶解土臭味素的直接生物降解以及过滤介质的生物再生,因为它从碳(解吸8,14)。
在这项研究中,我们调查了明尼苏达州圣保罗圣保罗地区水面(SPRWS)的全尺寸BAC过滤器的细菌群落动态。本研究的目的是了解这些全尺寸BAC过滤器的细菌群落结构,包括过滤器到过滤器的变异性及其相关的季节动态。使用PCR扩增的16S rRNA基因的V6区域的Illumina测序追踪细菌群落组成。这些结果表明,在BAC过滤器上生长的细菌群落是高度多样化的,但在所有过滤器和全年保持一些关键人群。最常见的种系是来自Nitrospira属通常认为是氧化亚硝酸盐的化石营养生物。奇怪的是,在Illumina谱中发现了很少已知的氨氧化细菌(AOB;即亚硝化单胞菌属)。然而,随后的定量实时PCR(qPCR)分析检测到大量的amoA基因与Illumina谱中的Nitrospira样群体的分数更一致。这种amoA比率之间的差异通过qPCR检测的16S rRNA基因和Illumina谱中已知AOB的比例表明这些细菌群落含有先前未鉴定的氨氧化微生物。我们推测AOB和亚硝酸盐氧化细菌(NOB)在这些BAC过滤器中占主导地位的原因是用氯碱化的成品水反冲过滤器,这种反应可以释放0.5到1.0毫克/升的氨(作为N)。颗粒活性炭(GAC)介质(15)。
SPRWS为居住在明尼苏达州圣保罗市和附近几个社区的近420,000人提供饮用水。该设施每天可处理1.44亿加仑,但目前平均每天生产4500万加仑。密西西比河是SPRWS的主要水源,尽管河水在进入设施之前经过三个不同的湖泊(Lake Pleasant湖,Sucker湖和Lake Vadnais湖)。处理过程包括一系列单元操作,包括凝结(硫酸铝),软化(添加石灰),絮凝,沉淀,过滤和消毒(氯胺化)。过滤系统包括24个独立的过滤床,每个过滤床在10厘米的沙子顶部含有约1米的GAC。每周对每个滤床进行一次反洗,以除去在GAC介质上随时间累积的颗粒。反冲洗过程始于空气冲刷(流速,25米3 / h),然后以每平方英尺5.5至13.8加仑每分钟的速度迫使水通过过滤器。
SPRWS人员收集用于化学分析的水样作为常规监测的一部分。在处理设施的入口处收集原水样品; 在水进入分配系统之前,从透明井中收集完成的水样。本研究中的所有化学分析均由SPRWS人员根据EPA方法350.1(氨),EPA方法353.2(亚硝酸盐/硝酸盐)和标准方法5310B(总有机碳[TOC])(16)进行。用于氨分析的方法还量化氯胺中含有的氮。
使用清洁和消毒的1升瓶从BAC过滤器的顶层收集样品,并立即运送到明尼苏达大学(<4小时)。将三份子样品(~0.5g [湿重])浸入裂解缓冲液(5%SDS,10mM NaPO 4,pH 8)中,然后在FastPrep机器(MP Biomedical,Solon,OH)中剧烈摇动30秒。使用Spin Kit for Soil(MP Biomedical)根据制造商的说明提取基因组DNA。将基因组DNA保存在-20℃直至需要。
如前所述(17)对每种DNA提取物进行靶向16S rRNA的V6区域的一式三份PCR 。简而言之,PCR引物是五种经过修饰的967F引物的混合物,这些引物已知可扩增细菌和古细菌(18),与1046R引物偶联,并在5'末端连接六碱基多重识别条形码(19))。最初使用2%琼脂糖凝胶筛选PCR产物,并使用QIAquick凝胶提取试剂盒(Qiagen,Valencia,CA)按照制造商的说明纯化。将纯化的PCR产物以相等的浓度合并,并在明尼苏达大学基因组学中心(UMGC)的MiSeq台式测序仪(Illumina,Inc.,San Diego,CA)上用作配对末端序列分析(2×150bp)的模板。 )。将PCR产物合并成四组20个单独的样品; 根据制造商的说明书进行文库制备和测序。
使用Eppendorf Mastercycler EP Realplex热循环仪(Eppendorf,Westbury,NY)进行定量实时PCR(qPCR)以定量16S rRNA基因(总细菌生物量的量度)(20)和两种不同的amoA基因进化枝(特异性)氨氧化菌及氨氧化古细菌,分别地)(21,22)。每个qPCR运行包括在95℃下初始变性10分钟,然后进行40个循环的95℃变性15秒,以及在靶特定温度下退火/延伸1分钟。25μl反应混合物含有12.5μl具有6-羧基-X-罗丹明(ROX)的iTaq SYBR green Supermix(Bio-Rad,Hercules,CA),25μg牛血清白蛋白(Roche Applied Science,Indianapolis,IN),优化的正向和反向引物数量,以及指定体积的模板DNA(通常为0.5μl)。根据经验优化每个样品的模板DNA的精确体积和浓度,以产生最低检测限,同时最小化PCR的抑制。本研究中使用的所有PCR引物的汇总表包含在补充材料中(参见数据集S1)。
基于使用已知量的模板DNA产生的标准曲线计算未知样品中靶DNA的量。通过PCR扩增来自阳性对照的基因,然后连接到StrataClone(Stratagene,Santa Clara,CA)或pGEM-T Easy(Promega,Madison,WI)克隆载体并转化到大肠杆菌 JM109中来制备qPCR的标准。使用QIAprep Spin Miniprep试剂盒纯化质粒。通过用Hoechst 33258染料染色来定量质粒DNA,并使用小牛胸腺DNA作为标准在TD-700荧光计(Turner Designs,Sunnyvale,CA)上测量。制备质粒DNA的系列稀释液并在每次qPCR期间运行以产生标准曲线(r 2> 0.99)。监测扩增效率以确保以相似的效率扩增样品和标准品(扩增效率为80至105%,取决于测定)。产生熔解曲线并分析以验证未发生非特异性扩增。
如上所述扩增来自两个BAC滤膜样品的amoA基因片段用于qPCR,纯化,连接到pGEM-T Easy克隆载体中,转化到大肠杆菌 JM109中,并铺板到补充有40μgml -1氨苄青霉素的LB琼脂平板上。 。随机挑选菌落,以便可以如上所述提取和纯化质粒。然后使用提取的质粒作为模板进行核苷酸序列分析,使用M13F和M13R作为测序引物。双向序列信息用于产生共有序列。通过使用ClustalW(23)最佳比对核苷酸序列产生系统发生树,然后使用邻接法推断系统发育距离(24)使用DNAMAN ver。7软件(Lynnon Corporation,Quebec,Canada)。参考序列从GenBank数据库获得(25)。
使用mothur处理和分析Illumina MiSeq序列数据(26)。筛选序列的质量; 在条形码引物中含有多于一个错配的序列,长度小于50bp或大于125bp,并且除去具有不明确碱基的序列或长于8bp的均聚物。此外,如果在15bp的滑动窗口中其MiSeq定义的平均质量分数小于25(Q25 =不正确的碱基调用的0.3%),则除去序列。资格序列按样本分类; 从序列读数中修剪引物和条形码。序列与SILVA细菌16S rRNA数据库对齐,UCHIME算法用于检测可能的嵌合序列,这些序列也从数据集中删除(27)。将Illumina序列聚类成操作分类单位(OTU),截止值为97%序列同一性。使用RDP数据库版本9(28)获得每个OTU的代表性序列的分类学信息。对社区概况进行非度量多维尺度缩放(nMDS),随机修剪到相同数量的序列以避免采样偏差。Shannon多样性指数,以及Chao1和基于丰度的覆盖估计(ACE)丰富度估计(29),是根据调整的和完整的OTU数据集计算的。
核苷酸序列可通过NCBI数据库以BioProject登录号PRJNA261440获得。
2007年,在SPRWS安装了20多个单独的BAC过滤装置,以改善成品水的味道和气味,取代以前使用的传统双介质过滤器(无烟煤和沙子)。安装BAC过滤器后的5年期间,与之前的5年期间相比,客户投诉减少了近90%(安装前,每年120±40次投诉;安装后,每年投诉13±4次) 。
在整个研究期间,跟踪了四个单独的BAC过滤器(过滤器6,9,17和20)去除总有机碳(TOC)的能力(图1A)。这些BAC过滤器去除了TOC的5%至15%,在夏季(5月至9月)和冬季月份具有相似的去除效率; 本研究中的原水温度范围为5°C(2011年3月)至28°C(2011年8月)。还跟踪了原水和成品水中的氨和亚硝酸盐/硝酸盐含量(注意用于检测游离氨的方法也检测了氯胺中的氮含量)(图1B)。原水中的氨水平通常等于或低于检测限(<0.1 mg升-1作为N),而亚硝酸盐/硝酸盐浓度范围为0.2至0.6mg升-1,为N(算术平均值= 0.38mg升-1,为N)。成品水中亚硝酸盐/硝酸盐含量没有显着高于原水中的(双尾t检验; P = 0.5)(算术平均值= 0.43 mg升-1,为N)。成品水中含有氯胺(3.5±0.2 mg升-1,为Cl 2),有可能在腐烂时释放出大量的氨(15),通常> 0.6 mg L -1 这种氯化成品水用于反冲洗BAC过滤器,从而为BAC过滤器提供潜在的氨源。
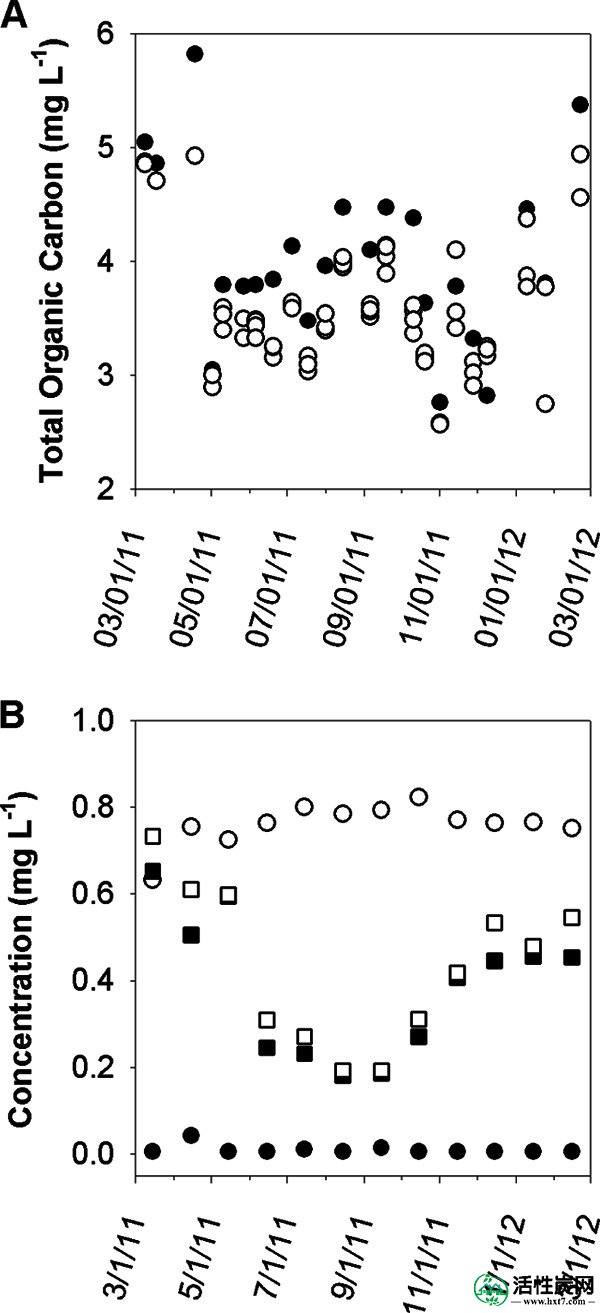
在本研究期间,SPRWS水处理设施的相关水质参数。(A)进入BAC过滤器(实心圆圈)的水和离开过滤器6,9,17和20的水(空心圆)中的总有机碳浓度。(B)原水(封闭符号)和完全处理过的水(空心符号)中氨(圆圈)和亚硝酸盐/硝酸盐(正方形)的浓度。
生成PCR扩增的16S rRNA基因片段的Illumina MiSeq谱,以允许在特定日期对细菌群落组成进行过滤 - 过滤比较以及遵循细菌群落动态。共获得430万个高质量DNA序列,每个序列的序列数为14,923至115,179(平均值53,700;标准差[SD],19,800)。在将序列聚类成> 97%序列同一性的阈值后,检测到总共4,674个不同的操作分类单位(OTU)。
BAC过滤器支持的细菌群落在研究它们的整年中令人惊讶地多样化(表1)。在将每个Illumina MiSeq谱随机调整至14,923个序列的大小(即,使所有谱图具有相同的读数)之后,在每个谱中检测到675至940个不同的OTU(平均值,824个OTU; SD,77) OTU),其中Chao1估计丰富度在1,200和1,800个OTU之间(平均值,1,480个OTU; SD,192个OTU)和ACE估计丰度在1,520和2,570个OTU之间(平均值,1950 OTU; SD,350 OTU)。香农多样性指数在整个日历年和过滤器与过滤器之间基本一致,范围从4.4到4.9(平均值,4.7;标准差,0.2)。奇怪的是,当Illumina MiSeq配置文件被分析而没有修剪到相同数量的序列时(参见补充材料中的数据集S5),检测到的OTU数量通常高于随机调整的配置文件的Chao1丰富度估计值。相比之下,修剪的轮廓的ACE丰富度估计通常与未修剪的轮廓中检测到的OTU的数量相匹配,这表明该估计方法在预测这些Illumina MiSeq轮廓的丰富度方面更准确。在修剪和未修剪的轮廓之间的Shannon多样性计算几乎没有差异,这表明修剪的Illumina MiSeq轮廓包含足够的序列深度以获得Shannon多样性指数的可靠估计,与先前的研究一致(表明这种估算方法在预测这些Illumina MiSeq谱的丰富度方面更准确。在修剪和未修剪的轮廓之间的Shannon多样性计算几乎没有差异,这表明修剪的Illumina MiSeq轮廓包含足够的序列深度以获得Shannon多样性指数的可靠估计,与先前的研究一致(表明这种估算方法在预测这些Illumina MiSeq谱的丰富度方面更准确。在修剪和未修剪的轮廓之间的Shannon多样性计算几乎没有差异,这表明修剪的Illumina MiSeq轮廓包含足够的序列深度以获得Shannon多样性指数的可靠估计,与先前的研究一致(30)。
通过PCR扩增的16S rRNA基因片段的 Illumina MiSeq分析测量的全尺寸BAC过滤器中观察到的OTU的数量,预测的OTU(通过Chao1和ACE估计)和细菌群落的Shannon指数
| 过滤 | 日期(月b日) |
OTU的数量
|
香农指数 | ||
|---|---|---|---|---|---|
| 观测到的 |
估计预测:
|
||||
| 超1 | 高手 | ||||
| 2 | 7月11日 | 834±23 | 1,486±100 | 1,929±155 | 4.7±0.1 |
| 3 | 3月11日 | 939±15 | 1,715±109 | 2,418±92 | 4.8±0.1 |
| 5月11日 | 886±89 | 1,635±248 | 2,347±446 | 4.7±0.1 | |
| 7月11日 | 860±64 | 1,583±204 | 2,178±313 | 4.7±0.1 | |
| 8月11日 | 835±97 | 1,447±244 | 1,833±488 | 4.9±0.1 | |
| 11月11日 | 791±60 | 1,422±167 | 1,806±323 | 4.6±0.03 | |
| 12月11日 | 752±35 | 1,274±64 | 1,544±71 | 4.6±0.04 | |
| 2月12日 | 759±26 | 1,356±96 | 1,929±156 | 4.4±0.2 | |
| 五 | 7月11日 | 813±29 | 1,441±105 | 1,825±223 | 4.8±0.04 |
| 6 | 3月11日 | 931±30 | 1,662±89 | 2,239±92 | 4.8±0.1 |
| 5月11日 | 917±37 | 1,784±86 | 2,567±110 | 4.7±0.2 | |
| 7月11日 | 842±21 | 1,558±141 | 2,077±134 | 4.6±0.2 | |
| 8月11日 | 779±8 | 1,339±62 | 1,590±18 | 4.8±0.02 | |
| 11月11日 | 814±33 | 1,416±101 | 1,741±241 | 4.7±0.06 | |
| 12月11日 | 793±22 | 1,364±96 | 1,736±172 | 4.7±0.04 | |
| 2月12日 | 751±12 | 1,377±58 | 1,802±67 | 4.4±0.04 | |
| 13 | 3月11日 | 917±11 | 1,733±188 | 2,397±300 | 4.5±0.05 |
| 5月11日 | 930±36 | 1,748±86 | 2,406±68 | 4.6±0.04 | |
| 7月11日 | 818±44 | 1,442±59 | 1,881±183 | 4.9±0.07 | |
| 8月11日 | 917 | 1,624 | 2,198 | 4.6 | |
| 10月11日 | 821±24 | 1,433±113 | 1,780±161 | 4.7±0.05 | |
| 11月11日 | 772±63 | 1,290±167 | 1,553±161 | 4.7±0.06 | |
| 12月11日 | 740±56 | 1,224±133 | 1,524±215 | 4.6±0.1 | |
| 2月12日 | 755±10 | 1,454±74 | 1,952±61 | 4.4±0.09 | |
| 17 | 7月11日 | 828±23 | 1,518±112 | 2,101±307 | 4.8±0.03 |
| 20 | 7月11日 | 826±9 | 1,458±21 | 1,959±116 | 4.8±0.09 |
| 22 | 7月11日 | 677±55 | 1,193±105 | 1,545±243 | 4.6±0.02 |
BAC过滤器的细菌群落组成在时间变异性和过滤器到过滤器变异性方面表现出相对较小的差异(图2)。三个BAC滤波器的细菌群落的组合物(过滤器3,6,和13)进行了超过12个月(在七个不同的场合监测图2A到TOC)。C)。这三个BAC过滤器中的每一个都是动态的,因为每个样本日期与其他每个样本日期相比显示出独特的社区概况。同样,这三个细菌群落在各个样本日期之间通常存在显着差异。为了进一步解决过滤器到过滤器的可变性问题,2011年7月对四个额外BAC过滤器(过滤器2,5,17和20)的细菌群落进行了分析(图2D); 除了过滤器2和3之外,这些过滤器社区中的每一个在该日期彼此显着不同。
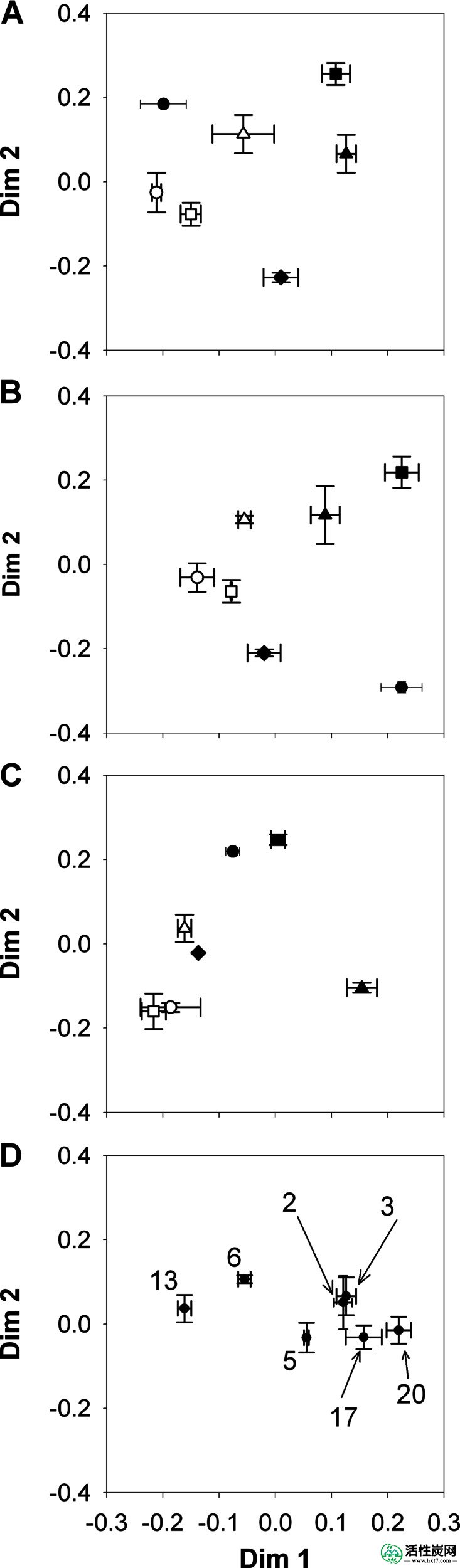
通过SPRWS的BAC过滤器中16S rRNA基因片段的PCR-Illumina谱的非度量多维比例(nMDS)测定的细菌群落结构的排序图。(A)过滤器3中的细菌群落动态。(B)过滤器6中的细菌群落动态。(C)过滤器13中的细菌群落动态。(D)2011年7月1日不同过滤器中细菌群落结构的比较; 不同的过滤器由数字表示。●,2011年3月15日; ■,2011年5月31日; ▲,2011年7月1日; ◆,2011年8月11日; ○,2011年11月22日; □,2011年12月15日; △,2012年2月14日。数据显示为三次重复曲线的算术平均值(±1标准偏差)。
相比之下,细菌群落中个体种群的变异随着时间的推移和过滤器之间的相对较小(图3)。所有样本中最主要的种群是Nitrospira sp。,占总社区的13%至21%。这个最显着的OTU也进行了人工分析,显示出对Nitrospira japonica菌株J1(登录号NR_114396)的98%序列同一性(63个核苷酸中的62个)(31)。虽然其他一些细菌种群的调节程度较大,但在12个月内,社区内15种最具优势的细菌种群数量不超过13倍(Variovorax sp。)(图3A)。在任何给定时间,从过滤器到过滤器的细菌群落组成变化较小,因为在8个不同过滤器中,群落中15个最主要的细菌群体的数量变化不超过8倍(也是 Variovorax sp。) 2011年7月(图3B)。
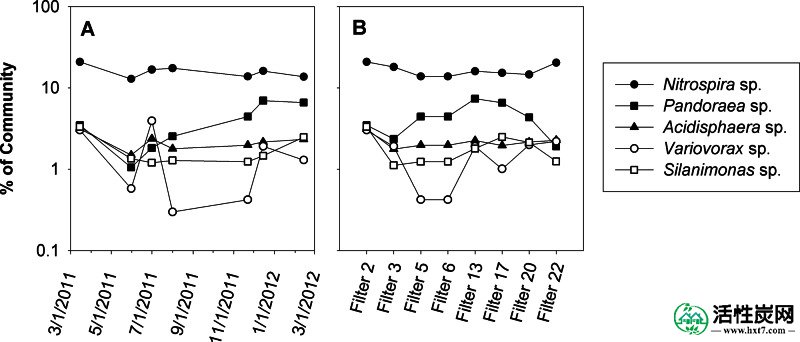
(A)PCR-Illumina谱中SPRWS过滤器3中几种最主要的细菌群体的动态。(B)2011年7月1日不同过滤器中代表性的单个细菌群体的比较。所有日期的所有细菌谱的PCR-Illumina数据包括在补充材料中。
的存在硝化菌,亚硝酸盐氧化菌(NOB)(32,33),因为细菌群落(> 16%),对这些BAC过滤器增长最突出的成员激起了我们的好奇心,因为很少(~0.07%)在Illumina MiSeq谱中检测到AOB(即亚硝化单胞菌的顺序)。此外,所有检测到的AOB都是Nitrosospira spp。没有Nitrosococcus spp。,厌氧铵 - 氧化细菌,或氨氧化古菌检测到(AOA)。尽管源水中存在高浓度的亚硝酸盐(SPRWS用来表征其水的分析方法无法区分亚硝酸盐和硝酸盐),但亚硝酸盐在环境中通常是不稳定的,并且不太可能存在大量的亚硝酸盐。亚硝酸盐将在源水中维持较长时间。因此,我们惊讶地发现大量的NOB没有AOB和/或AOA的可比较量(即,在一个数量级内)。
为了进一步检查BAC过滤器中的AOB和AOA,针对来自已知AOB和AOA的amoA基因片段进行qPCR 。没有AMOA在BAC过滤社区被检测到来自AOA基因。相反,使用qPCR技术检测到大量的细菌amoA基因,该技术先前报道为对亚硝化单胞菌寡聚样amoA基因具有特异性(即,我们的样品中也可能存在其他amoA基因类型)。当细菌amoA基因片段通过每个样品中16S rRNA基因的总数(也通过qPCR定量)标准化时,amoA的量基因大大超过Illumina MiSeq谱中已知AOB的分数,并且与Nitrospira spp 的分数更紧密地对齐。在Illumina MiSeq配置文件中(图4)。来自过滤器3,6和13(n= 15)的Illumina MiSeq曲线中所有AOB的分数的log 10值的算术平均值为-3.2,标准偏差为0.3。相反,通过qPCR检测的amoA与16S rRNA基因(n = 15)的log 10比率的算术平均值为-1.8,标准偏差为0.5。log 10数量的这个差异相当于25倍的差异; 双尾t这些分布的测试证实这些方法的差异具有统计学意义(P <0.0001)。
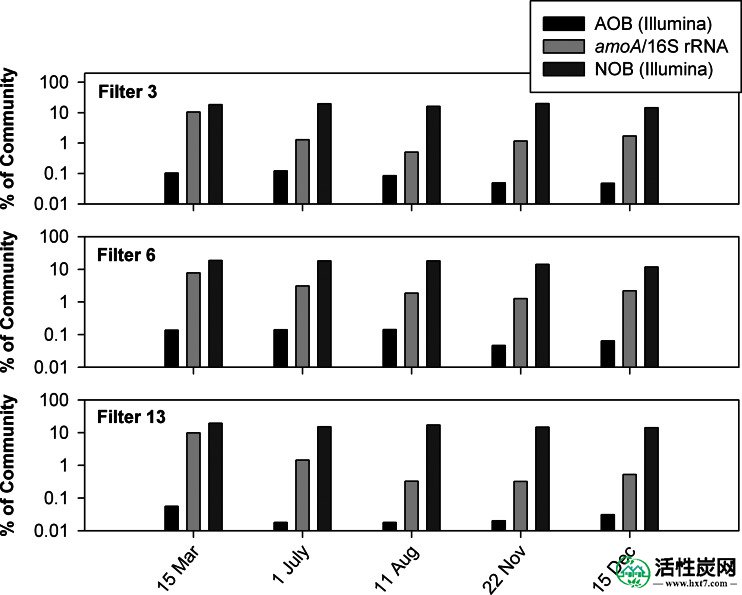
比较三种SPRWS BAC过滤器中氨氧化和亚硝酸盐氧化细菌的相对量。AOB(Illumina)和NOB(Illumina)的值分别代表Illumina谱中发现的已知AOB和Nitrospira序列的分数除以该谱中序列的总数。所述AMOA /的16S rRNA代表的比率AMOA如通过定量实时PCR(见数据的补充材料设置S2至S4)测定于16SrRNA的基因。
因为定量PCR引物设计为特异于亚硝化单胞oligotropha样AMOA基因片段(34),但没有亚硝化单胞oligotropha样在Illumina的MiSeq轮廓检测16S rRNA基因片段,AMOA基因片段从在从过滤器6收集的样品扩增2011年8月和2011年12月,克隆并测序以确认扩增的amoA基因片段是真实的。对总共34个克隆进行测序,产生两个独特的DNA序列,其中一个检测到31次,另一个检测到三次。两个克隆的序列与amoA紧密聚集从充分表征的AOB分离株中鉴定出的基因(图5)。这些克隆中更突出的细胞与亚硝化单胞菌(Nitrosomonas sp。)最为密切。菌株Is79A3,而较不突出的克隆合理地聚集在Nitrosomonas oligotropha附近。
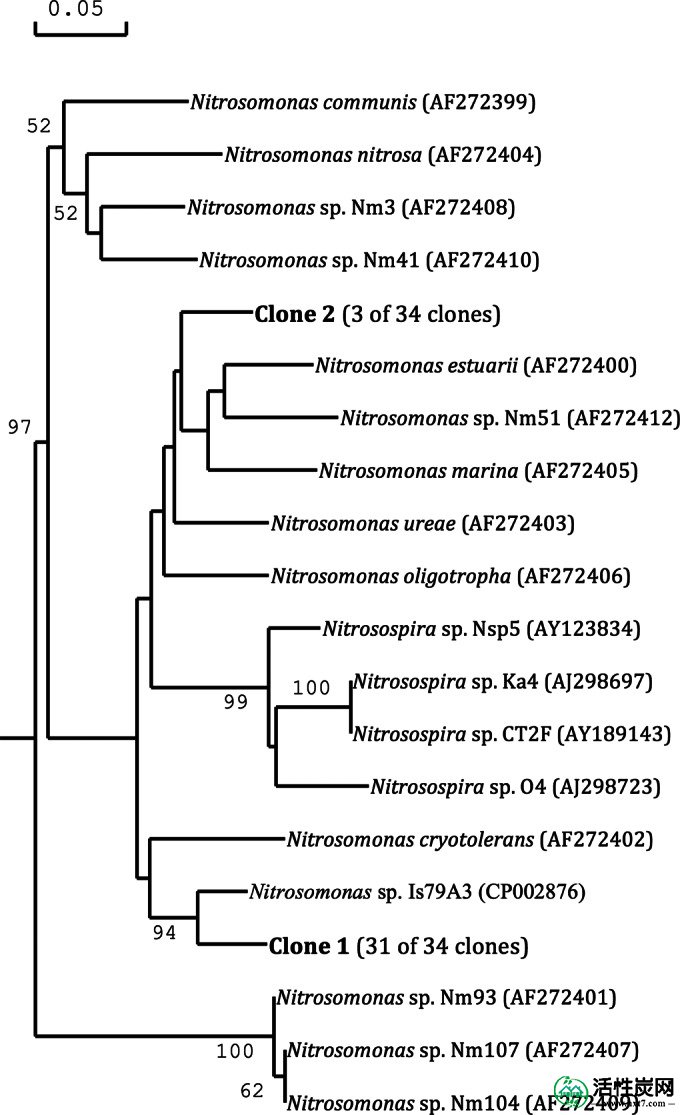
树状图显示在该研究中获得的amoA基因片段序列(164个核苷酸)的系统发育相关性与来自已知氨氧化细菌的amoA基因序列相比较。参考序列的GenBank登录号显示在括号中。
本研究表明,SPRWS用于生产饮用水的全尺寸BAC过滤器含有高度多样化和稳定的细菌群落。从生态观点来看,这种高度的细菌的多样性应该导致更有效的营养物处理,改进的生态系统的稳定性,和耐工艺失常(35,- 41)。从实际角度来看,该技术的应用导致水消费者的味道和气味投诉显着减少。这并不奇怪,因为传统的生物活性过滤器(即没有GAC培养基)可以有效地去除许多有关的微污染物(42))。因此,将细菌介导的转化与大量吸附能力相结合,如同这些BAC过滤器一样,对于微污染物去除应该更具吸引力。
我们的研究还表明,以前无特征的氨氧化微生物在这些BAC过滤器中很突出。支持这一主张的最佳证据是Illumina MiSeq谱中AOB数量与“ 亚硝化单胞菌特异性” amoA基因数量之间的差异为25倍(34),这表明能够进行氨氧化的基因是存在的数量显着高于先前已知含有这些基因的生物。此外,因为本研究中使用的PCR引物对已知amoA基因序列的一小部分具有高度特异性,实际差异可能甚至大于25倍。amoA的相对数量每个细胞的16S rRNA基因也可以正面或负面地影响这种差异。先前的研究人员假设2个AMOA基因和3.6 RRN每个基因组(操纵子34,43)分别AOB和所有的细菌,虽然目前还不清楚这些假设将如何有效的是在这里分析的BAC过滤器的微生物。相比之下,Baptista等人。(44)最近观察到通过qPCR靶向amoA基因获得的定量之间的良好一致性,使用先前显示对非特异性扩增敏感的PCR引物(34),以及针对该命令的荧光原位杂交(FISH)Nitrosomonadales。在同一项研究中,这两种方法都超过了qPCR靶向亚硝化单胞菌的16S rRNA基因所获得的数量级数量级或更多(32)。不幸的是,我们无法在本研究中针对Nitrosomonadales的目标进行FISH 。
因为所有基于PCR的测定可能偏向于或针对特定的微生物群体,所以考虑许多替代解释来协调Illumina MiSeq谱中的AOB数量和通过qPCR测量的amoA基因的量。一种可能性是AOA,的数量显著已报道在以往的研究上BAC滤波器(存在45,46); 然而,针对已知AOA 的amoA基因的qPCR 是阴性的(注意我们已经在明尼阿波利斯市的中试规模BAC过滤器中使用这种方法来成功量化amoA来自AOA的基因[未发表的结果])。接下来,靶向16S rRNA基因的V6区域和/或Illumina MiSeq分析的PCR可能以某种方式偏向已知的AOB。然而,对来自亚硝化单胞菌属的公开可用的16S rRNA序列的分析证实了与本研究中使用的PCR引物100%的序列同一性。另外,随后将未加工的Illumina MiSeq数据中人工放置的亚硝化单胞菌序列鉴定为AOB序列。此外,从SPRWS的中试规模(数据未显示)和全尺寸BAC过滤器收集的其他样品获得了类似的结果,目标是16S rRNA基因的V3区域(参见补充材料中的数据集S6和S7),表明PCR偏向于订单的AOB亚硝化单胞菌不太可能,因为针对16S rRNA基因的不同区域的两个PCR方案对同一系统发育组具有相似的偏倚是非常不可能的。最后,在我们的分析的DNA提取/纯化步骤中也可能存在对AOB的偏见。然而,我们以前使用这种DNA提取程序与靶向16S rRNA基因的V3区域(没有Illumina衔接子和条形码)的PCR引物组合以表征其中亚硝化单胞菌属的硝化富集培养物。包含多达25%至70%的细菌群落(47),再次表明对亚硝化单胞菌的AOB的分析偏差不太可能。
支持声称新型氨氧化生物存在于BAC过滤器中的第二个(但更弱)证据是Illumina MiSeq谱中NOB与AOB的比例极高(NOB / AOB比率为225)。假设氨氧化微生物产生亚硝酸盐作为NOB的唯一电子供体,并且两种生物都是化石营养型自养生物,那么AOB与NOB的比例应该反映这两种生态位的各自代谢反应的能量可用性。 。热力学计算表明,从氨氧化成亚硝酸盐(ΔG °'= -275 kJ mol -1)可获得的能量大于亚硝酸盐氧化成硝酸盐的能量(Δ)G °'= -76kJ mol -1)。相比之下,许多先前的研究人员报道,使用多种不同方法,NOB群体在微生物群落中的AOB群体大于AOB群体。例如,据报道,使用qPCR ,NOB / AOB比率从低至2.5(21)至高达195(34); 使用FISH,其他研究人员已经报道NOB / AOB比率为10〜30(范围48,49)。在克隆的,几乎完整的16S rRNA基因库中,这个比例是来自饮用水生物过滤器的50(50)。总之,热力学计算表明AOB应该超过细菌群落中的NOB,这与经验证据相矛盾,其中NOB通常比AOB更突出。一种替代解释是NOB能够通过混合营养生活方式吸收简单的有机化合物而超过AOB,这最近由Nitrospira sp 的基因组提出。(33)。无论如何,需要进一步研究以更好地理解AOB和NOB在复杂细菌群落中的关系和作用。
Nitrospira spp 的优势。在Illumina群落中,qPCR检测到的细菌amoA基因丰度是出乎意料的,因为源水含有可忽略不计的氨量(图1B))。因此,我们假设在这些BAC过滤器中驱动硝化微生物生长的氨的最终来源来自有机氮(未测量),其在处理过程中通过氨化转化为氨。此外,我们推测用氯化成品水反洗的做法,这可以在腐烂时释放0.5至1.0毫克/升的氨,也有助于氨氧化微生物和NOB在细菌群落中占优势。利用反冲洗流量(5.7升m -2 h -1)和频率(每7天~20分钟)并且假设AOB的典型细胞产量,我们计算出反洗可能是造成AOB增加的原因,AOB占总细菌群落的0.2%(计算未显示)。这具有实际意义,因为BAC过滤器中的AOB可以为饮用水分配系统提供连续的硝化细菌来源(51),可能导致硝化事件,从而导致不可接受水平的亚硝酸盐积累(52))以及有意维护的残留氯胺的衰变以防止分配系统内病原生物的再生。另一种可能会减少BAC过滤器中硝化细菌数量的策略是在消毒之前(即而不是之后)用过滤水反冲洗这些过滤器。
总之,SPRWS使用的BAC过滤器保持了非常多样化的细菌群落,能够显着改善处理过的水的味道和气味。这些细菌群落在一年中保持稳定,并且以Nitrospira sp 为主。还有尚未鉴定的氨氧化微生物。先前未鉴定的氨氧化微生物的存在并非没有优先权,因为培养实际上是鉴定新型氨氧化微生物的先决条件,并且迄今为止大多数原核生物都抵抗微生物培养基上的分离(53)。即便如此,我们对氨氧化微生物的了解在过去的二十年中已大大扩展,包括可在低pH下生长的AOB(54)),厌氧氨氧化细菌(55),氨氧化古菌(56),甚至建议可以存在一种可以将氨氧化成硝酸盐的单一生物(57)。最近,其他研究人员还提出,以前未被认识的氨氧化微生物在实验室规模的废水生物反应器中表现突出,在低溶解氧浓度下进行硝化(58)。这里分析的细菌群落含有新型氨氧化微生物的可能性无疑得益于新的DNA测序技术(即Illumina MiSeq)的应用,该技术允许更细致地分析细菌群落组成,特别是“次要”人口占总社区不到1%。