发布日期:2018-11-07 13:38 来源:活性炭网 作者:活性炭网 浏览数:
抽象 我们从基于吸附剂的修复现场试验中收集了受多环芳烃(PAHs)影响的城市土壤样品,以解决2%粉状(PAC)或颗粒状(GAC)活性炭修正对土壤微生物学和污染物生物降解的不良副作
目前正在研究添加活性炭(AC)作为原位技术来修复受污染的沉积物和土壤。1在好氧土壤中应用的一个关键考虑因素是AC对内在微生物群落结构和功能的长期影响,包括生物降解污染物的潜力。加入AC到土壤将减少疏水性有机化合物(肝卵圆细胞)的土壤孔隙水浓度2将由AC(图被吸附和强结合(图1,1,方法I)。在AC的微孔束缚污染物变得不易进入用于通过土壤栖息生物biouptake 3 - 5和植物4,6与弱结合或溶解的污染物相比。AC修改可以由此减少肝卵圆细胞从土壤基质转移到陆地食物链和也减少植物毒性7(图(图1,1,方法III),加入AC的土壤也将减少肝卵圆细胞浸出8(图(图1,1,流程iv)和挥发风险5(图(图1,1,过程ii)。然而,AC对HOC的强结合将降低HOCs对具有代谢这些化合物能力的微生物的可用性。潜在的后果可能是HOC降解物从主要的土壤微生物群落中消失和/或HOCs代谢的下调有利于其他碳底物。在具有丰富的肝卵圆细胞降解微生物的土壤中,这可以通过生物降解(图损害污染衰减(图1,1,流程v)中。
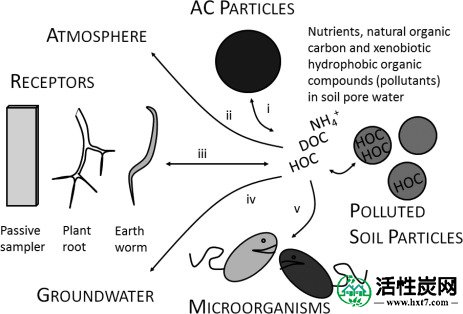
在活性炭(AC)改良土壤中影响疏水性有机化合物(HOCs)命运的各种方法的链接的图示。(i)AC修正增加了HOC的吸附并降低了溶解在孔隙水中的HOC的浓度。减少孔隙水HOC浓度典型地暗示(ⅱ)少肝卵圆细胞挥发到大气中,5(iii)通过蚯蚓和植物,肝卵圆细胞的biouptake少7,21和(iv)小于肝卵圆细胞浸出到地下水。8降低的HOC孔隙水浓度也可能改变土壤微生物群落结构和功能,包括(v)HOCs的生物降解。AC对其他基质的二次影响,如溶解的有机碳和营养素浓度,8 土壤的润湿性和物理土壤结构也可能改变土壤微生物群落的结构和功能。
有报道称强吸附剂修正对污染物生物降解过程有不同的影响。在一方面,除了AC到污染土壤显示出降低掺加14 Ç菲代谢至14 CO 2在实验室分批实验,9和Karapanagioti等。图10显示缓慢的吸附动力学限制了含有煤颗粒的沉积物浆液中的加标菲生物降解。另一方面,Vasilyeva等人。7表明AC有助于克服多氯联苯对微生物的毒性,Payne等人。11还报道了添加AC对沉积物中的PCB脱氯有轻微的刺激作用。Bushnaf等人。12报道,生物炭改性土壤中单芳烃含量的降低导致了线性,支链和环状烷烃的更大的生物降解,并且在含有和不含2%生物炭的砂质土壤中,总石油烃蒸气降解具有可比性。在AC修正与生物刺激和生物强化的直接比较中,Hale等。13发现2%AC添加比生物刺激或生物强化更有效,进一步降低了泰恩河沉积物中已经强烈隔离的多环芳烃(PAH)污染的可用性。
本文的目的是研究AC修正对主要细菌群落结构的影响及其功能,重点是PAHs的生物降解。据我们所知,这是有史以来第一次调查AC修正对现实田间条件下土壤微生物群落的长期影响。我们报告了修复PAH污染的城市土壤中颗粒状或粉状活性炭(GAC和PAC)后三年内主要细菌群落结构的变化。对于第3年收集的样本,我们使用分子微生物学和化学方法更详细地比较了细菌群落及其功能。
Hale等人报道了基于AC的土壤修复的田间试验的细节。8蒸渗仪实验中使用的土壤在挪威德拉门的一个建筑工地进行挖掘,使用2%湿土重量PAC的挖掘机铲斗混合(SilCarbon TH90,平均粒径20μm,80%<45μm)或GAC(SilCarbon0.3-0.8,300-800μm粒度),并嵌入室外渗透仪中,表面积为25 m 2,深度为2.5至3 m。土壤最初的总有机碳含量为2.50±0.04%干重,修正的土壤含有预期的AC剂量,尽管具有相当大的变异性,GAC为2.0±1.0%或PAC为2.4±1.9%。8在AC修正土壤后,6个月后和3年后立即进行两系列用于微生物分析的土壤样品,并在-20℃下冷冻,加入和不加入无水乙醇1:1(v / v) )。对于3年样品,将额外的土壤样品储存在4℃的冷室中,不添加乙醇用于批次实验。
将储存在乙醇中的第3年样品中的10μL加入到990μL过滤器灭菌的磷酸盐缓冲盐水(PBS,Oxoid)中,并通过在100×浓缩的二甲基亚砜中添加50μL的SYBR Gold核酸染色剂来染色细胞( DMSO)(Invitrogen Ltd.,Paisley,UK),用箔包裹并在室温下孵育30分钟,之后使用无菌Millipore过滤器支架和0.2mm孔径的黑色聚碳酸酯过滤器(直径25)过滤它们。 mm; Millipore)。将滤膜转移到含有一滴Citifluor(Citifluor Ltd.,Canterbury,United Kingdom)抗褪色剂的玻璃显微镜载玻片上,以帮助粘附到载玻片上。将另一滴Citifluor置于过滤器的顶部,并将盖玻片小心地放置在制剂的顶部。使用Olympus BX40落射荧光显微镜,通过油浸物镜(100×放大倍数)下的直接计数测定总细菌数; 使用稀释产生20个随机选择的视野,所述稀释产生30至300个具有清晰轮廓和有限细胞形状的荧光细胞。使用每次处理测量的三次重复的平均值的标准偏差估计样品之间的误差。通过考虑过滤膜的数量和视野面积,膜的面积和原始样品稀释因子来确定每g土壤的细胞数。使用稀释产生20个随机选择的视野,所述稀释产生30至300个具有清晰轮廓和有限细胞形状的荧光细胞。使用每次处理测量的三次重复的平均值的标准偏差估计样品之间的误差。通过考虑过滤膜的数量和视野面积,膜的面积和原始样品稀释因子来确定每g土壤的细胞数。使用稀释产生20个随机选择的视野,所述稀释产生30至300个具有清晰轮廓和有限细胞形状的荧光细胞。使用每次处理测量的三次重复的平均值的标准偏差估计样品之间的误差。通过考虑过滤膜的数量和视野面积,膜的面积和原始样品稀释因子来确定每g土壤的细胞数。14
通过监测含有15g湿重未经修正的,PAC修正的或GAC修正的土壤的50-mL卷曲顶部小瓶中的CO 2浓度来测量第3年样品的土壤呼吸。对于每种土壤类型,在室温(20℃)下监测重复批次4天。在与具有HP-PLOT-Q毛细管柱的Fisons MD800 MS连接的Fisons 8060 GC上进行CO 2的 GC-MS分析。
进行指纹方法,变性梯度凝胶电泳(DGGE),以确定实验处理中主要细菌群落之间的相似性和差异,并鉴定这些群落的选定成员。从在储存(不含乙醇)土壤样品的0.5g(湿重)等分试样中提取总细菌DNA,在蒸渗仪实验开始后0和6个月和3年时取得。使用Fast DNA Soil Kit(Qbiogene)进行DNA提取,纯化步骤,由Griffiths等人的方法修改,15在核酸提取之前加入,以防止已知抑制PCR扩增的化合物如腐殖酸和粘土矿物的共提取。简而言之,预处理包括使用十六烷基三甲基溴化铵(CTAB,Sigma-Aldrich)提取缓冲液和苯酚/氯仿/异戊醇(25:24:1,Sigma-Aldrich)从土壤基质中提取核酸,然后使用苯酚用氯仿/异戊醇(24:1,Sigma-Aldrich)除去。引物2和3,靶向细菌,使用PCR扩增的细菌的16S rRNA基因片段的V3区域,如先前通过Muyzer等人,描述16和如先前所描述的PCR产物通过DGGE分析。13使用图像分析软件BioNumerics(Applied Maths NV,St.Martens-Latem,Belgium)对DGGE图像进行标准化和解释。引物6(Primer-E Ltd.,Plymouth,UK)用于使用Pearson积矩相关系数和相似性分析(ANOSIM)进行聚类分析。
从凝胶上切下显性DGGE条带,使用引物2/3进行PCR扩增,纯化并使用ABI prism Big Dye Terminator Cycle Sequencing Ready反应试剂盒和ABI Prism用引物2或3(3.2pmol /μes/μL)测序。如前所述的377 DNA测序仪(Applied Biosystems,USA)。13使用BLAST算法将核酸序列数据库项目(RDP10)和GenBank数据库的序列进行比较,以确定最接近的匹配序列同一性。
特定环羟基化双加氧酶α亚单位的丰度(RHD α)基因通过实时PCR在每个样品中使用以下引物组测量:(ⅰ)P6B(正向(F):5'-TGGCGAACTCGTGTCGGCAC-3';反向(r):5'-CGTCCAGRCAACCGAADAYC -3'),靶向pdoA2 / phdA进化枝,其主要包括分枝杆菌属物种并且具有M.vanbaalenii作为参考菌株; (ii)P4(f:5'-CCGGAGACTTCCTGACGAC-3'; r:5'-GCASACGAAYCGACGGGT-3')靶向ebdA1 / etbA1 / akbA1b进化枝,其仅包括红球菌属物种并且具有R.jostii RHA1作为参考菌株; (iii)P7B(f:5'-CACBTGCAGCTAYCACG -3'; r:5'-CATGTGGTCCATGTAGAAC -3')靶向aipbA1 / bphA1进化枝,主要包括红球菌和一些假单胞菌属,特别是包括几种红细胞内红细胞。株。使用C1000热循环仪iCycler和软件版本1.6(BioRad CFX Manager)在BioRad CFX96(Hercules,CA)上进行实时PCR实验。10μL反应混合物含有3μL模板DNA(或过滤的无菌分子生物学水,Sigma-Aldrich,作为阴性对照)和15pmol的每种引物,其中SsoFast EvaGreen Supermix用于CFX96(Biorad Laboratories Ltd.)。在扩增中使用以下温度曲线:步骤1加热至98℃(2分钟),然后进行40个循环,在98℃下变性2秒,在引物特异性退火温度下5秒(P6B为58℃) ,P4为60°C,P7B为59°C)。在实时PCR结束时,进行解链曲线作为最终步骤,其包括在0期间测量SYBR Green信号强度。在65至95°C之间每10秒钟升温2°C。荧光与温度变化的对应曲线显示每个扩增子在其特定解链温度下的单峰。对于每组引物,从由含有克隆的靶序列的质粒制备的标准DNA获得校准曲线。使用Nanodrop 1000分光光度计(Thermo Scientific)定量质粒DNA浓度。根据质粒大小,插入物长度和假设每个碱基对的分子量为660Da计算标准质粒的拷贝数。制备标准DNA的储备溶液,浓度为10 从含有克隆的靶序列的质粒制备的标准DNA获得校准曲线。使用Nanodrop 1000分光光度计(Thermo Scientific)定量质粒DNA浓度。根据质粒大小,插入物长度和假设每个碱基对的分子量为660Da计算标准质粒的拷贝数。制备标准DNA的储备溶液,浓度为10 从含有克隆的靶序列的质粒制备的标准DNA获得校准曲线。使用Nanodrop 1000分光光度计(Thermo Scientific)定量质粒DNA浓度。根据质粒大小,插入物长度和假设每个碱基对的分子量为660Da计算标准质粒的拷贝数。制备标准DNA的储备溶液,浓度为10质粒μL -1的9个拷贝。对于校准曲线,通过稀释储备溶液制备范围为10 9至10 1个靶基因拷贝μL - 1的 DNA标准品。所有标准曲线在9个数量级(10 9至101μL - 1)内呈线性(R 2 > 0.99))基因拷贝数。每个样品一式三份进行,每个实验重复至少两次。在每次运行中,通过比较qPCR产物的熔解曲线和琼脂糖分析与从模式生物中提取的DNA(阳性对照)获得的精确度来确认土壤样品中靶基因检测的准确性,以避免环境样本中的错误检测。
建立了一系列批量实验来研究被动采样器中可利用的PAHs的摄取。13被动采样器能够从被污染的水中被动地积聚HOC,直到建立分区平衡。通过被动采样吸收污染物潜在地也可用于摄取临界受体如植物和蚯蚓(图(图1,1,方法III)。为了研究单独吸附(图的影响(图1,1,方法I )相比吸附和生物降解的组合效果(图(图1,1关于污染物可用性的过程i和v),在添加和不添加叠氮化物的情况下建立批次,其抑制PAH生物降解。17,18微观世界包括以下内容:5克(湿重)未改良土壤,PAC修正土壤或GAC修正土壤和40毫升水。样品的水含量为16至21%湿重。对于每种土壤类型,在有和没有1g / L叠氮化钠(Sigma Aldrich)的情况下建立三个批次。每批加入0.15±0.01克清洁聚乙烯(PE)被动采样器。空白对照平行进行,由含有PE取样器和含有和不含叠氮化钠的水的三份批次组成。将批次用棉球塞住,并在轨道振动器(IKA labortechnik,德国)上以100rpm的速度在一个二级黑暗容器中混合,所述二级暗容器由纸板箱组成,其中一些孔被冲入盖子中用于通风。3周后除去PE取样器,并在10mL 50:50v:v己烷/丙酮中萃取两次,持续24小时。d 10 -菲,83±6%的d 10 -芘,为92±4%d 12 -benzanthracene,和94±8%d 12 -benzperyl。
使用安捷伦6850气相色谱仪(DB-XLB柱长30 m,内径25 mm,膜厚1μm)进行GC-MS分析,使用硅胶柱顶部硫酸钠进行样品净化,并与Agilent 5973质谱仪连接如Hale等人所述。8
在修正后3年,观察到总细胞计数的统计学显着性差异,GAC修正的土壤具有1.8×10 10个细胞/克干土重的最高细胞数,比未修复的土壤高2.5倍。最低细胞计数(图(Figure2a)。2一个)。这些值是在较高范围为最典型的土壤类型(例如,(19,20))。土壤呼吸速率也为2.6和统计学显著因子2.7更高的GAC-修正土壤相比,未修改的和PAC-修正土壤(图(Figure2b)。2 B)。土壤呼吸导致了CO的线性增加2批次顶部空间浓度在整个4天监测期间对所有土壤(- [R 2个值> 0.98),与土壤呼吸可以被量化为0.19±0.02,0.18±0.01,和0.49±0.03微克CO 2每克每干土的未修正,PAC和GAC修正土壤的小时数。这些观察结果与GAC修正土壤中更好的植物生长的实地观察一致,参见Jakob等。21土壤呼吸速率与另一项关于碳氢化合物污染场地的研究中发现的值相似。22这些结果表明AC修正总体上对有氧微生物活性没有害处,尽管先前已报道在AC修正土壤中增强了DOC结合。8
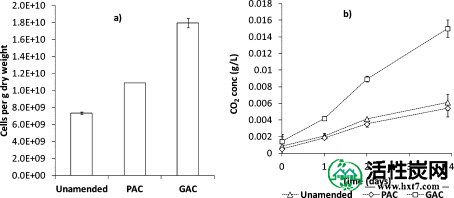
(a)3年后采集的土壤样品的总微生物细胞计数和(b)相同样品的土壤呼吸。误差线表示方法部分中定义的重复的标准偏差。不可见误差条小于图中的线或符号。
主要的土壤细菌群落结构,通过DGGE和0后修正和未修正的土壤的聚类分析,6和36个月评估(图(Figure3),3),表明所有各特定时间周期将样品紧密聚集来自同一时期的其他样本,无论治疗如何。这表明主要的细菌群落结构随时间的变化比对PAC或GAC修正的响应更强(ANOSIM测试时间组之间的差异,全球R = 0.852,p <0.01;对于治疗之间的差异,全球R = -0.317,p= 0.97)。总体而言,主要的细菌群落,通过皮尔逊积矩相关系数确定之间的相似性,大于65%(图(Figure3b)。3 b)中。从相同的时间点和治疗重复DNA样品显示出更高的相似性(> 77整体%,平均相似系数> 87%,图Figure3b),3b),表明良好的方法重现性。最丰富的微生物物种是土壤中碳和养分循环的重要驱动因素。本研究中观察到的AC修正和未修复土壤中主要细菌群落结构的相似性相当高,减轻了对AC修正的强烈副作用的担忧。这些发现与沉积物无脊椎动物场社区调查同意其中AC修正案通常只有在群落结构和多样性,弱影响23,24虽然在一个领域研究中观察到物种丰富清晰的效果。24
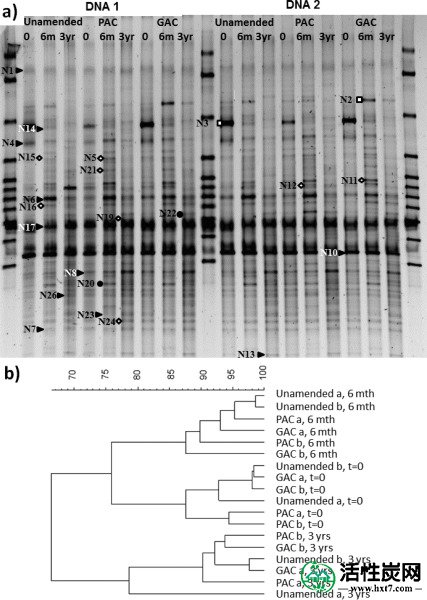
(a)DGGE凝胶显示切除条带的位置,与三类环境细菌相对应(右实心三角形=放线菌;◊=变形菌;□=拟杆菌;●=未知)。最匹配的序列在支持信息的表S1中报告。在零时间,6个月和3年时,从未修饰的和AC修正的土壤(PAC,GAC)中提取DNA两次(DNA1和DNA2)。(b)树状图显示了DGGE检测的治疗的不同群落组成随时间的相似性(Pearson积矩相关系数)的聚类分析。
使用基本局部比对搜索工具(BLAST)鉴定与测序的DGGE条带最接近的匹配邻居,并使用核糖体数据库项目(RDP 10)中的分类器工具确认分类。二十三的26最主要类群测序并分为三个主要的类群,拟杆菌,放线菌,和变形菌(由不同的符号识别,图Figure33和表S1 支持信息)窝藏已知常见环境细菌在有机物分解中起重要作用。25 - 28三种最丰富和普遍的分类群(带N6,N10,N17和分别)属于顺序Micrococcinaea的内放线菌门,其最近的邻居被确定为一个Curtobacterium属,节杆菌,和微杆菌物种虽然具有变化的相似性(89-99% ; 支持信息中的表S1 )。这些细菌是常见的土壤居民。几个主要丰富(强带)分类群的最近匹配邻居是有意义的,因为据报道在这些分类群的成员中降解PAHs具有广泛的能力(例如,(29)):N7带与分枝杆菌 96%相同SP。和92%类似多功能PAH降解M. vanbaalenii PYR-1,30,31带N8是类似于通用的PAH降解95%红球菌josttii应变RHA1,32带N16 95%是相同的一个未经培养鞘氨醇藻。和带N23是类似于物种的菌株99%红串红球菌,是已知的成员降解联苯和异丙基苯(例如,参考文献(33,34))。这些基团通常存在于所有土壤(例如,(35)),但似乎在污染土壤频繁支配多个(例如,(36,37)),尽管据我们所知,获得与含有分枝菌酸的放线菌(例如,红球菌和分枝杆菌)匹配的序列的这种优势是不寻常的。这些条带存在在从未经修正的,PAC-细菌群落的所有次,和GAC-修正土壤,但它们的相对强度随时间变化(见表表1)。1)。条带的相对强度可以提供社区中这些分类群的比例丰度的粗略代表。这些条带在第3年的相对强度平均强于研究开始时的相对强度,但在处理之间相当,这表明这些推定的PAH降解类群在土壤中具有相似的相对丰度。该R. jostii RHA-1样序列(N8带)在研究开始和结束之间显示出特别显着的强度增加,其中从主要细菌群落的约3%增加到近10%,如AC修正的相对强度所表示的和未经修正的土壤。
|
N7 DGGE 分枝杆菌样序列
|
N8 DGGE Rhodococcus RHA1样序列
|
N23 DGGE Rhodococcus erythropolis- like序列
|
||||||
|---|---|---|---|---|---|---|---|---|
|
相对带强度%
|
相对带强度%
|
相对带强度%
|
||||||
| 泥 | 时间 | 样品 | 平均 | 平均 | 平均 | |||
| 未修改 | 0 | DNA1 | 1.87 | 1.52±0.50 | 2.93 | 2.72±0.30 | 1.59 | 1.43±0.22 |
| DNA2 | 1.16 | 2.51 | 1.28 | |||||
| 6个月 | DNA1 | 4.50 | 4.46±0.05 | 3.26 | 3.30±0.06 | 1.84 | 1.87±0.04 | |
| DNA2 | 4.42 | 3.34 | 1.90 | |||||
| 3年 | DNA1 | 3.00 | 2.96±0.06 | 8.36 | 8.92±0.80 | 2.21 | 2.35±0.20 | |
| DNA2 | 2.92 | 9.48 | 2.50 | |||||
| PAC | 0 | DNA1 | 3.48 | 3.15±0.46 | 4.65 | 4.26±0.55 | 2.22 | 2.16±0.08 |
| DNA2 | 2.83 | 3.87 | 2.11 | |||||
| 6个月 | DNA1 | 3.42 | 3.04±0.54 | 3.34 | 3.33±0.004 | 1.94 | 2.00±0.08 | |
| DNA2 | 2.65 | 3.33 | 2.06 | |||||
| 3年 | DNA1 | 3.25 | 3.49±0.34 | 9.37 | 9.26±0.16 | 3.32 | 3.03±0.40 | |
| DNA2 | 3.73 | 9.14 | 2.74 | |||||
| GAC | 0 | DNA1 | 1.96 | 1.84±0.17 | 3.53 | 3.19±0.50 | 1.66 | 1.57±0.12 |
| DNA2 | 1.71 | 2.85 | 1.48 | |||||
| 6个月 | DNA1 | 4.11 | 2.64±2.08 | 3.33 | 2.71±0.89 | 2.11 | 1.82±0.40 | |
| DNA2 | 1.17 | 2.08 | 1.54 | |||||
| 3年 | DNA1 | 3.73 | 3.34±0.55 | 8.55 | 7.90±0.92 | 2.77 | 2.67±0.15 | |
| DNA2 | 2.96 | 7.25 | 2.57 | |||||
实时PCR是用于定量功能性PAH环羟基化双加氧酶的有价值的工具。38,39双加氧酶系统的分子氧的两个原子与芳香环添加为较低分子量的多环芳烃的需氧降解的第一步,和双加氧酶基因的定量因此在PAH生物修复可能性进行评估的兴趣。40,41的RHD α报道在表基因拷贝数表22表明,由基准表示的那些红球菌 RHA1物种(从这里通称为ETBA RHA1 -RHD α)显示了最高的浓度(约107 -10 8克-1),而那些用a表示红串红球菌参考物种(从这里通称为IPDA Reryth -RHD α)表现出最低的浓度(约10 5 -10 6克-1)对于每治疗。那些由附图表示结核分枝vanbaalenii PYR1物种(从这里通称为pdoA2 PYR1 -RHD α)具有中间浓度(〜10 6 -10 7克-1)。这些推定的芳环降解细菌(即,RHD α如果假定每个细菌细胞含有一个基因拷贝,则分析的细菌占总细菌群体的0.003和1%之间。这些值类似于Cebron等人报道的绝对和相对丰度值。39使用定量PCR靶向受污染土壤中的一般双加氧酶基因群体,但高于仅针对特定双加氧酶基因群体的研究。42 - 44每个目标基因的浓度未修改土壤之间的数量级内下降,土壤修正与PAC和土壤与GAC修正。事实上,主要的ETBA RHA1 -RHD α基因群体在试验期间增加,并且与未修饰的渗透计相比在AC修正中更大程度地增加。这证实具有降解PAHs能力的细菌保留在PAC和GAC修正的土壤中,其水平与未修饰土壤中的种群相当。实时PCR方法测量基因数量,或降低PAHs的潜力,而不是基因表达,可能会被某些污染物降解剂下调。45换言之,而结果表明,以合成双加氧酶的能力是存在于从土壤样品中分离的所有的细菌群落,而不管AC修正,这并不一定意味着酶的活性合成。有趣的是,要注意的是有针对性的RHD之间的显著相关α基因丰度和由有密切的隶属关系的那些RHD的参考物种DGGE分析(相对强度×总细胞计数)中找到的那些分类群的浓度α基因(Pearson相关,- [R 2 = 0.83,P <0.01,Ñ = 9) 。然而,尽管有用于RHD的每个目标进化枝参考物种这些16S rRNA基因片段的密切联系α,所述相似性往往比通常用于物种水平那些截止值低(例如,92-96%)(97% ),它不能自信地认为,这些类群必然包含RHD α基因,即使它们被认为是相似的类群。
|
模型生物
|
分枝杆菌vanbaalenii
|
Rhodococcus jostii RHA1
|
红球菌(Rhodococcus erythropolis)
|
|||||
|---|---|---|---|---|---|---|---|---|
|
靶基因
|
phdA,pdoA2
|
ebdA1,etbA1,akbA1b
|
ipbA1,bphA1
|
|||||
| 泥 | 时间 | 样品 | 基因拷贝n / g湿土 | 平均 | 基因拷贝n / g湿土 | 平均 | 基因拷贝n / g湿土 | 平均 |
| 未修改 | 0 | DNA1 | 1.18±0.13×107 | 13.9±1.5×106 | 3.76±0.35×107 | 37.2±5.1×10 6 | 1.95±0.70×106 | 2.39±0.48×106 |
| DNA2 | 1.60±0.17×107 | 3.69±0.68×107 | 2.83±0.23×106 | |||||
| 6个月 | DNA1 | 3.11±0.42×106 | 3.1±0.3×106 | 8.45±0.04×107 | 77.8±4.0×10 6 | 2.85±0.33×106 | 2.77±0.55×106 | |
| DNA2 | 3.11±0.15×106 | 7.12±0.74×107 | 2.69±0.78×106 | |||||
| 3年了 | DNA1 | 3.12±0.44×106 | 3.3±0.3×106 | 1.20±0.09×108 | 119.0±10.0×106 | 7.85±0.85×105 | 0.71±0.06×106 | |
| DNA2 | 3.45±0.21×106 | 1.17±0.10×108 | 6.43±0.40×105 | |||||
| PAC | 0 | DNA1 | 1.98±0.31×107 | 18.3±1.8×106 | 4.43±0.54×107 | 44.0±4.5×10 6 | 2.72±0.26×106 | 2.49±0.39×106 |
| DNA2 | 1.69±0.04×107 | 4.38±0.36×107 | 2.26±0.52×106 | |||||
| 6个月 | DNA1 | 3.60±0.20×106 | 3.6±0.5×106 | 7.75±0.99×107 | 77.8±8.8×10 6 | 6.34±0.80×106 | 6.79±0.61×106 | |
| DNA2 | 3.51±0.70×106 | 7.80±0.77×107 | 7.24±0.42×106 | |||||
| 3年了 | DNA1 | 4.47±0.90×106 | 4.9±0.6×106 | 1.79±0.17×108 | 175.0±10.0×106 | 1.55±0.08×106 | 1.31±0.05×106 | |
| DNA2 | 5.22±0.26×106 | 1.71±0.04×108 | 1.07±0.03×106 | |||||
| GAC | 0 | DNA1 | 9.48±0.40×106 | 10.4±0.6×106 | 3.37±0.12×107 | 34.7±4.4×10 6 | 1.37±0.30×106 | 1.57±0.25×106 |
| DNA2 | 1.13±0.90×107 | 3.57±0.76×107 | 1.78±0.22×106 | |||||
| 6个月 | DNA1 | 2.56±0.81×106 | 2.5±0.6×106 | 8.43±0.13×107 | 79.6±8.5×10 6 | 2.50±0.16×106 | 2.37±0.22×106 | |
| DNA2 | 2.45±0.38×106 | 7.49±0.43×107 | 2.23±0.28×106 | |||||
| 3年了 | DNA1 | 5.29±0.90×106 | 5.7±0.8×106 | 1.72±0.23×108 | 177.0±17.0×106 | 4.79±0.32×105 | 0.67±0.20×106 | |
| DNA2 | 6.12±0.63×106 | 1.82±0.13×108 | 8.76±0.07×105 | |||||
假设每细胞310 FG的碳含量46和一个ETBA RHA1 -RHD α每个细胞的基因拷贝,对应于表的基因拷贝数的生物质碳表22将是每克干土40和70之间微克,这大于固相PAH浓度,远大于每克土壤低于1 ng的自由溶解PAH浓度。8比较表明,含有双加氧酶基因的微生物利用了其他主要的碳底物,因此可以在有和没有AC修正的土壤中长期生长和持续存在。该研究结果缓解了AC增强的PAH结合可能导致AC修正土壤中推定的PAH降解物丰度显着降低的担忧。
PE无源取样器用于含有和不含杀生物剂,叠氮化钠的分批系统,并显示对AC的吸附和生物降解均影响了在蒸渗仪实验开始3年后土壤样品中PAHs的可用性(图(图4)。4)。被动采样器采用的多环芳烃被认为可被生物和植物吸收,并且往往提供与生态毒性评估相比更好的与土壤总浓度的相关性。47的PAC-修正无菌土壤测量的高的标准偏差可能是由于在PAC的分布小尺度非均质8,48或PAH的污染土壤中,49因为一个PE采样器比其他两个重复采样具有更高的PAH摄取。然而,对于与叠氮化钠,其通常加入到批来说明吸附的效果而已,土壤泥浆2有一个在PAH摄取统计学显著减少由PE采样器用于AC修正的土壤,对于减少57%与未修正的土壤相比,GAC修正案和PAC修正案减少69%(t检验,单尾,GAC的p <0.01和PAC的p <0.05)。在比较含有和不含叠氮化钠的土壤浆料时,PE被动采样器对未经修正和GAC修正土壤的PAH吸收的统计显着降低75%和46%,说明了生物降解的影响(吨测试,单尾,p <0.01和未经修正的p <0.05 GAC)。PAH化合物模式也表明生物降解; 吸收较小的两环和三环PAH化合物,往往更容易生物降解,40与未加入的土壤相比平均降低99%,与叠氮化钠相比没有(表S2在支持信息中))。总体而言,没有叠氮化钠的PAC修正土壤中PAH摄取量最低,我们的观察结果显示,尽管AC修正土壤和未修复土壤中PAH含量低,但PAH生物降解活跃,缓解了AC修正可能导致PAH代谢下调的担忧。然而,如果不添加杀菌剂,AC修正案的益处就不太明显了。对于不含叠氮化钠的土壤浆料,与未改良土壤相比,PAC改良土壤仅减少44%具有统计学意义(t检验,单尾,p<0.05)。因此,为了在现场条件下对AC修正的长期效益进行实际评估,应对无菌土壤和活土壤进行实验室试验,以解释由于生物降解可能在对照土壤中发生的污染减弱。
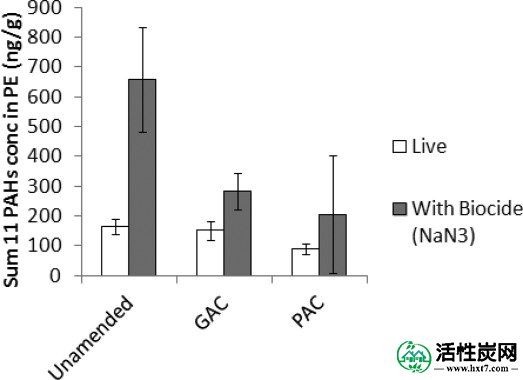
在未经修饰的PAC-缓慢混合浆液中嵌入3周的被动采样器吸收PAH。和GAC修改的土壤在3年后取样。显示了16种USEPA PAH化合物中的11种(PE中浓度高于分析检测限的那些:ace烯,ace,芴,菲,蒽,荧蒽,芘,苯并(a)蒽,chrysene,indeno(123 cd) )芘和苯并(ghi)per)。杀生物剂抑制含有1g / L叠氮化钠的批次。
把这些结果放入上下文中,我们在图比较Figure55个游离的水性PAH浓度Ç 瓦特从在聚乙烯的PAH的浓度估计,Ç PE,用PE-水分配系数ķ PE和关系Ç 瓦特 = Ç PE / ķ PE,50与由Hale等人报道游离的水性PAH的浓度。8它们来源于被动聚甲醛(POM)采样器嵌入式场渗透仪。由于温度和混合方式的差异,原位和非原位评估不具有完全可比性,这在实验室条件下更为优化,因此倾向于促进污染物质量转移和生物降解。48尽管如此,综合数据似乎显示,在3年修复试验期间,AC修正和未修复土壤中游离水中PAH浓度持续下降,最终降至欧盟制定的欧盟饮用水指导值以下的极低水平。所有系统中的指令200/60 / EC。总体趋势是所有衰减过程的综合影响,包括PAH吸附,生物降解,化学反应,浸出和挥发。51随着时间的推移,AC修正系统中加速污染衰减与自然污染衰减之间的差异变得越来越小,并且当自然衰减使现有的PAH池耗尽到与AC修正相同的范围时,可能最终收敛。根据我们的结果,可用PAHs的生物降解是游离水性PAH浓度降低的重要因素,即使在田间实验期间每克未处理的干燥土壤21的总 PAH浓度为23±15μg 也没有显着改变。8 AC修正的益处,例如减少蚯蚓和植物对PAH的吸收21和减少PAH浸出,8 与内在衰减机制的工作相比,需要评估和AC修正成本,这也可能长期降低风险,特别是在污染物降解微生物丰富的土壤中。
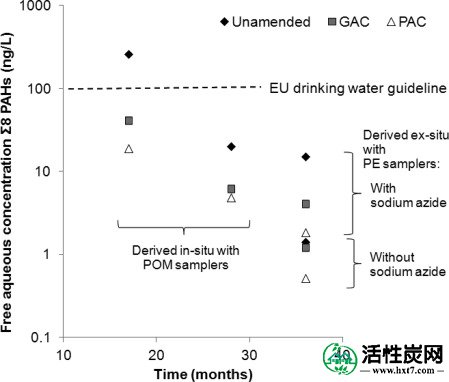
比较在非原生批次试验中第3年测量的PE采样器浓度的游离水性PAH浓度与Hale等人报道的现场数据,8来自于在17和28个月后嵌入渗透仪中的POM取样器。开始实验。显示了16种USEPA PAHs的化合物的总和,这两种化合物可以在两个研究中定量:菲,蒽,荧蒽,芘,苯并(a)蒽,chrysene,茚(123cd)芘和苯并(ghi)per。